신기술
수처리 과정서 항생제 내성 유전자 분해 예측 모델 개발
이윤호 GIST 교수팀, 미 워싱턴 대학과 공동 연구…분해 기작·속도 규명
유전자로 구성된 환경·보건 오염물질의 효율적 제거 위한 지식기반 제공
수처리 과정에서 항생제 내성 유전자의 분해 정도를 정확하게 예측할 수 있는 모델이 탄생했다. 광주과학기술원(GIST)은 이윤호 지구·환경공학부 교수팀이 미국 워싱턴 대학과 공동 연구를 통해 수처리 소독 과정에서 항생제 내성 유전자의 분해 기작과 속도를 규명했다고 지난 2월 18일 밝혔다.
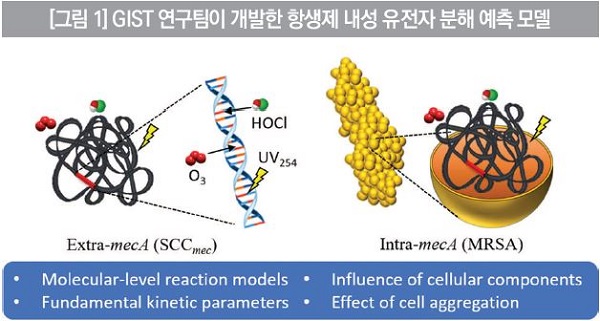
공공 보건 측면에서 항생제 내성 유전자는 새로운 유형의 환경 오염물로 인식되고 있다. 오랜 항생제 오남용으로 생활하수나 병원, 축산 폐수 등에서 항생제에 저항을 갖는 내성균들이 발생했고, 이들은 항생제 내성의 정보가 저장된 유전자를 사람과 위험한 병원성 미생물에게 전파했다.
하수나 폐수의 적절한 소독을 통해 항생제 내성균·내성유전자를 처리함으로써 항생제 내성 전파를 효과적으로 차단시킬 수 있다. 하지만 항생제 내성 유전자가 수처리 소독제와 얼마나 빠르게, 어떤 형태로 반응하는지는 알려지 않아 수처리 소독 방법이 충분한지에 관한 연구가 필요한 상황이었다.
연구팀은 대표적 항생제 내성균인 ‘MRSA’와 MRSA가 항생제에 대해 내성을 가지게 하는 ‘mecA’ 유전자를 대표 균주·유전자로 두고 연구를 진행했다. 연구 결과, 각각의 소독제들은 mecA 유전자와 고유의 반응 특성을 보였으며 염소는 ‘두단계 반응 모델’을, 오존은 ‘이차 반응 모델’, 자외선은 ‘중합체 형성 및 풀림 가역 반응 모델’로 mecA 유전자가 분해되는 속도를 효과적으로 기술할 수 있음을 확인했다.
이번 연구에서 개발한 반응 모델은 내성 유전자 염기서열 정보를 활용해 소독 처리에 따른 내성유전자 제거 정도를 예측할 수 있게 해준다는 점에서 중요성을 갖는다. 개발된 모델을 활용한 결과, 일반적 하수처리 소독공정 조건에서 mecA의 처리 효율은 99.9% 이상으로 나타났다. 다만 mecA 유전자가 MRSA 내부에 존재할 경우에는 분해 속도가 훨씬 느렸는데 이는 MRSA 균이 집합체를 형성해 소독제와의 반응을 저해하기 때문이다. 이는 실제 수처리 조건에서 내성유전자 처리 효율이 낮을 수 있음을 시사하며 관련 후속 연구가 필요한 상황이다.
이윤호 교수는 “이번 연구는 유전자로 구성된 새로운 환경 오염물질의 수처리 효율을 분자 수준의 반응 이해를 통해 체계적으로 접근한 새로운 시도”라며 “향후 다양한 항생제 내성 유전자와 더불어 최근 문제가 되고 있는 신종 바이러스에도 적용할 수 있을 것으로 기대된다”고 말했다.
한편 이번 연구는 미국 시애틀 워싱턴 대학의 마이크 도드(Mike C. Dodd) 교수팀과 공동 협력의 결과물로 GIST 박사과정의 최예균 학생이 6개월간 시애틀을 방문해 일부 실험을 수행했으며 한국연구재단 중견연구자 사업의 지원을 받아 수행됐다.
연구 성과는 환경과학 분야 국제학술지인 『환경과학 및 기술(Environmental Science and Technology)』 1월 26일자 온라인판에 게재됐다.
[『워터저널』 2021년 3월호에 게재]

